DreINT0145145 @ danRer10
Intron Retention
Gene
ENSDARG00000094612 | si:dkey-262g12.3
Description
si:dkey-262g12.3 [Source:ZFIN;Acc:ZDB-GENE-060503-255]
Coordinates
chr4:72140078-72145569:-
Coord C1 exon
chr4:72145423-72145569
Coord A exon
chr4:72140403-72145422
Coord C2 exon
chr4:72140078-72140402
Length
5020 bp
Sequences
Splice sites
5' ss Seq
CAGGTAAGG
5' ss Score
11.08
3' ss Seq
TGTTTTATATTTCTCTTTAGAGC
3' ss Score
10
Exon sequences
Seq C1 exon
GACAAAGTGTCCAAACACAGAGGAAAACTCCTGCTGAAGTGACTGATCTCCACACTGTTATAAAGATGGCGTTTATTAAAGAGGAGAGTGAAGACCTGAAGATTGAAGACACATTCACAATCAAACAGGAAGATCCTGGTCAAACAG
Seq A exon
GTAAGGGCACATTTTGAATAAATGTCTGCTTTTACAGCAGGTCACATTGTAAATATGGAAAGATACGTTTTTATTCCCTGCTGAAAAATTCAGCTTATACCAGCCTAGGCTGGTTGGCTGGTTTTAGCTGGCCGACCAGGCTGGTTTTAGAGGGGTTTTGGCCATTTCCAGGCTGGTTTCCAGTCAATTCCAGCCTGGTCTTAGCTAGTCAGGCTGGAAAATGACCAGCTTGACCAGCCTGGTTTAAGCTGGACATAGCTGGTTTTGGCTGGGCTCCCAGCCTGGCTAGGCTGGTCAAGCTGGTTTTAGCTGGTCATCTCCCAGCTAGACTAGCTAAGACCAGGCTGGAAATGACTGGAAATGGCCAAAACCCCTCTAAAACCAGCCTGGTTGGCCAGCTAAAACCAGCCAACCAGCCTAGGCTGGTTTAAGCTGGTTTTTTCAGTAGGGAATTAAGTTAAGAAACCGTTAAAAATGGTTTAAACTATCGAAAAAGATGCTAAAACCCTAATCGCTTCTAGTGAATTGTTTTTCCATTACAAAATTGTCACGTTTAAGATCTGTTTTCTTTAAAAATCTTTAATCTTCACTGTTTGTTAGATATACGACAAAACGTAGTAGTTCACACTACAGGACTTTAAACATCGACAGATCGCTGTGCCATTCGCACTACATGACTTGATCTTGTGTCTTTAAATCGTTGTGGTGTTCACACTACGCGACACTACGTAAATGATCCACTAGAGGGCGGTCACACACACTACATGATCTGACAACAACTCATTTGCCAGCAACTCACTCAAGCTACCAAACCTCAGCCAACGAAATTTTGAGGGAGTCGTCATCAGAAAAAAGCTGAACACTATCCACAGCCAATCCGCTTGTTAAGTGTGACGTCACGCGAAGTGGCTTCCGGGTCCAAGCACTCTATTCAACTGAATGGGGAGACTCATGAAATGGTAATAATAAACGTTTACAAAGCGATTTAATAGTTTCGAAAGTCACGTTCGCAATATATATATATATATATATATATATATATATATATATCCATGCCTAATATCCGATGGCCAGTCTATGAGCTTAAAGAGCTGTATGCACCTCTGAAAGGATGTAGTGGACTCAAACATGGCTCTAAATGTATCGACGCTGTTTATCATCCTTCATAAAGCACATAAACACTGTTATTTTCTTCATAAAATCCATGTTAGCCCTTTAGCTATGAAGCTAGAGTCACGGGGCAGACAGAAGCCCTGCCCATCACGCGAATCCGCGTCTGTTCTGAGGTGAATTTGACACGCGAATGAAGCGAGTAACCTCAAATGTTCATGCGACTATTTACGCGCAAATAGCGTGATTTATCCGCGCGTTCCGCGTGTGGTGTGAACACAGCATTATGCAGCAAGCCCAGAGATTGTTGTGAACACTATGATTTTATATACAATTAACTTTAATATGTGATAGGAATAAAACGTGATCATAAACGAATGATTTCTCCACTCAAATGAATGGCGGCTTGGACCCGGAAACAGTATTACATACGTCACCAACACCGGCCCTCAAGGAACAAGTTTGGTGATCCCCCGTTTTGAGGCATCTTTTGATAGAATAATGCTCAACCTACCCCCCAAAACCCCCAGCACGTAAGTGTCATTATCGCCACCTGTTGGTTCAGGCTGCCCTTAAAGGTTCAAGTTTTTAAAAAATTGTAACATATTTGTGTGAGTTGAGCATCATTTAAGACAACGTTAGCATCTGTCAGCTTTTAATGTGGGGAAAACTAGATAATTTTGAGTAGCTAATTTCATCATACGGGTTTAAAATAATGTTTGGGGTGGGATCAAAATCGGTGAAGTAGCGCAAAACTGCTAGTGGAGTGATGGCCTAATGCCACGGGAATCCTAATAATAAGCCAGGTTAGTGCAAGAGTGGAGTCATACCACAGAGTGGCACGACTGTGTTATGTAAGCTCTGTTTTTGGGACTTCAGCTTACTGTCTCCTTGCGAGTAGAGGTCTTCACATTGGACTCGAAACCTGTGCGTGTTTGGGTCTATATTTGAAGTGATCAGTCAGGTCCCAATATATAATATATTGCATAGGTCTCAAGCTCAATTCCAGGAGGGCCGCAGCTCTGCCAGTTTTGCTCCAATCCTAATCAAACAAAGCTGATAAAACCTATCAAGATGTCTAGCAGTCTTGGATTACCTTGATTAGCTAGATTAGGGTTGGGGAATTGAGTGTGAGACCTGTGATATATTGACTGTTTCTCAATTCCAGGAACGCAGAGAACGGACTTGCGTTCTTGTGAAGAGCGGTCTTGCCAGGTCACCTCGGAAGAGAGAACTCGATAGACCACGAGAACAGAGAACGCGTCATGTGAAGAATGAGATGCTGCGTTCTTCCTGATAGTCACGTGACCTTCACGTGTTTTTAATGGAAAATTATTTAAACATTACAGAATTCATACGATTTTTTTGTTTTTCCCGTTTTCAAAATATATACTTTGCATAAAGACCTTACAAATATACTTGGCACAATATAAATTAAACAGATTTCAATACGAATTTCAGCAAATAAACACCCTTAATGTGTTTATTCCTTTATTAAGATTTTCATATATTCGCCACTTAATTTAATGAAACTCCTGAGGTAAATAAGTTCTATCTCTGAACTTTAATAATAAACCTATACAAAATGCTGCGCTTCCCACCTCCAGTCGCTTCTGGGACTTCTAGAGCGAGTTTGGTGCTCAAGTCTGCATCGAAGCCTTCTCGATATCACAAAGACAAGAAGTTTCACGCGTCCTCCGTTCTTGAGTCTTGGTACCAAACTCTGGCAGTTGATGATGATGTTACACGAGGAGGCAAGATCGCTGAAGAATGCATATTGAGAAACAGCCATCAGCCCCTTTCACACATACACACCTTTCCGGAAAATTACCGTCAATTTTCCGGAAAGGTGTGTATGTGTGAACAGGCTCTTTTTGTAAATACCGGTAAATCCGTTCTGGCTATTTTCCGCAAAGAGAAGTTGTAAGATTACCGGTAATTTGCCGGAATGCTGCGCTGTGTGAACGCAAAAGGAAGATTGCCGGAAAGAGCGCGTGCACGTCTAGAACGTGCTGACGTGAGACGTCTGCGTTAGCCAATCAGAACAGTCAGAGGCATTCACGTCCACGCGGTTTATGAGAATAAAAGCCTTTGAAGATTTTTCCAGGCACATTTAGCTGCTAGAAATTAGTCAGATAACGTTTATATGTTCATCTTAATGCCAACCGTGTAATTAATTATCGATAAGATGCTTATGATAAGCCGTTGTTTGTTTACCTTCAAGCTTTGCGTGTGCCCATAAACAGTCTATGAGCGCCAGCACACACACATATCATGTACATCTGGACATGCGAAAGTGTTTCTCTATATGTTTTCATCGAAGTTGTTCACAATACTGATTCATCCACAGAGTTTGTGATATAGTCAAATGTTTACAAATACAAGCACAGCCGCTTAGAGGTCTTTTCTGGTGAATGATGTCAGAATTTACCGCTTTTTTGGAATGGATGTGTGAACGGTCTTTTCCGGAAAAATTCCGTAACGTCCTCGCCTCTGTGAACAGCTCTTTTTTGAATTTACCGATAAAGTCGTTCCGGAAATTTTCCATATATTTACCGGTATCACTGTGTGAAAGGGGCTATTGTGTCTGTGTGTAATACCAGAGTCTGAACTCTGGAATTACACCGCGTCAACCAGTGATCAGTGGTCGGCACTTTGGATGTGTTTTGTAACTTGTAGATGTAAGAAGGTAATAAAATGTTTAAAACACACAAGTGTGCACTACCTTAGCCCAAGGAAGCGGAGCTTGGATTCGTTTTCATGAGGTTAAATGTAATAATAATGCATCAGTTGTTGCCTTGGTAATGTAAGTGTAATGCATGCATTGATATGGATCAGATATATCAGCAAAAACGTGCTGTCCAATGATATCAGTGGGTTCATCTGAATGACGTTTCTTTTTTTTTTTTGTGGTAACATTACCAAAATATTTAAGACCTATCGATGCTGAATTCAGGTGTCACGGTGGTGCAGTGGGTAGCACGATCGCCTCACATCTCACAAGAAGGTCGCTTGTTCAAGCCCCGGCTGGGTCACTTGGCATTTCTGTGTGGAGTTTGCATGTTCTCCCCATGTTCCCATGGGTTTCCTCAGGGTGCCCCCACAGTCCAAACACATGGGCTTTAGGTGAATTGGGTACGCAAAATTGGCCGTAGTGTGTATGTGTGAATGCAAGAGTCTGTTTGTGTTTCCCAATGTTGGGATTTGGCTGGAAGGGCATCCTCTGCGTGAAACATGTGCTGGATAAGATGACGGTTCATTTCGCTGTGGCGACCCCAGATTAATAGAGGGACTAAGCCGAAAACCCTGCTGAAAAATCCAGCTTAAACCAGCCTAGGCTGTTTGGCTGGTTTTAGCTGGTCGACCAGCCTGGTTTTAGAGGGTTTTTGGCCATTTCCAGGCTGGTTTCCACCCATTTCCAGCCTGGTCCTAGCTGGGCAGGCTGGAAAATGACCAGCCAAATCCAGCTAAAACCAGCTCAACCAGCCTGGTTGAAGCTGGATATAGCTGGTTTTGGCTGGACTCCCAGCTTGGCTAGGCTGATCAAGTTGGTTTTAGCTGGTCATCCAGCCAAGCTAAGACCAGGCTGGAAATGGCTGGAAAGCAGCCTGGAAGTGGCCAAAACCCCTCTAAAATAAGGCTGGTCGACCAGCTAAAACCAGCCTAGGCTGGTTTAAGCTGGATTTTTTTAGCAGGGAAGAAAATGAATGAATAAATGAATGATTCTGAATTCAGGACTTTTTGAGGACCAAAGGCCATCCTGTATTAAGACACTCTTAGGTTATATGTAACGCCTCTTTTAGCATCTTAGTACTTGTAAGAGCTGTTGTTGGTTTTAATACTTGCATTCTTTCATGTTGGTTTTTACATGTGTTGCTTTTGGGTTAACCTGTTTTATATTTCTCTTTAG
Seq C2 exon
AGCTGACGATGCTGAAAGAAGAGACTCAAGAACTGAATGAAATGGAAGAGAAAGATCAAGATTTAAAGACCGTTGAAAAATCCACAAAAATAAAAAGGACTTCACCTCATAAAAGAGATCAGGAAACCAAATCTACTGGTAACTTTACCTGCAGTCAGTGTGGGAAATGTTTTGCTCAAAAACACAACCTTAGAGTCCACATGAGTGCTCACACCAGAGAGAAGCCTCACTTGTGTCCTGAGTGTGGAAAGAGTTTCAGACGGAAGCAATACCTTGCTGTTCACATGAGAATTCACACCGGAGAGAAGCCTTTCTCGTGTGATCA
VastDB Features
Vast-tools module Information
Secondary ID
ENSDARG00000094612:ENSDART00000136328:2
Average complexity
IR
Mappability confidence:
NA
Protein Impact
Alternative protein isoforms
No structure available
Features
Disorder rate (Iupred):
C1=0.714 A=NA C2=0.445
Domain overlap (PFAM):
C1:
PF0263410=FdhD-NarQ=PU(0.1=0.0)
A:
NA
C2:
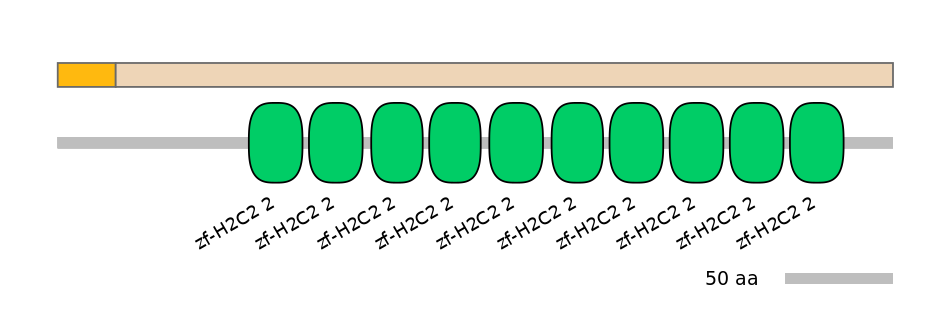
PF0263410=FdhD-NarQ=WD(100=91.7),PF097235=Zn-ribbon_8=WD(100=39.4),PF134651=zf-H2C2_2=WD(100=23.9),PF134651=zf-H2C2_2=WD(100=16.5)
Main Inclusion Isoform:
NA
Other Inclusion Isoforms:
NA
Associated events
Conservation
Mouse
(mm10)
No conservation detected
Mouse
(mm9)
No conservation detected
Chicken
(galGal4)
No conservation detected
Fruitfly
(dm6)
No conservation detected
Primers PCR
Suggestions for RT-PCR validation
F:
No suggested primer sequences
R:
No suggested primer sequences
Band lengths:
Functional annotations
There are 0 annotated functions for this event
GENOMIC CONTEXT[edit]
INCLUSION PATTERN[edit]