DreINT0068481 @ danRer10
Intron Retention
Gene
ENSDARG00000051768 | fhod1
Description
formin homology 2 domain containing 1 [Source:ZFIN;Acc:ZDB-GENE-110623-4]
Coordinates
chr7:34321049-34326407:+
Coord C1 exon
chr7:34321049-34321141
Coord A exon
chr7:34321142-34326270
Coord C2 exon
chr7:34326271-34326407
Length
5129 bp
Sequences
Splice sites
5' ss Seq
CAAGTAAGT
5' ss Score
10.08
3' ss Seq
TCTTCTCCGGTGCTTTACAGAGA
3' ss Score
7.8
Exon sequences
Seq C1 exon
AGCGTCTTTCTCAGAAACGGATTCTGCTCCGGAGACTCCAACTCAGACGACAGCAGATCAGCCCAGCCGGACAGTGGGAAAGAGAGTCTTCAA
Seq A exon
GTAAGTCATCAACTCTGATAGGAGGAAACAGGGGAGAAGGCAAAGGGAAAAGATGTGAGAGGAGGTGAAGAAATTGGAGGAGGGAATATTTTTGGGAAGGAGTTGAACTTCTTGAATGAGCTAAAGTGCCGCAAGCTGAAGTGAACTGCTAGAGCTGCGATTGAAACATTTCACAATTGGACTTGATTCATTGACTTTTGAATTATAGGACCATAGGTAAGTCTTCACTTGCTGATGGCTCCAGTAAGTGCTAACTGGCATGTTAATTATGAAAAAATCAGGAATCAATAGACTGAGGATTCATTTAGTTTTTTCTCTTCTAAAGCTTGATCTCAGGTGTGTTTCTTACTTTTATCTTCTTATACACAGAAATTCTTTATTCTGTGCTTAGCTCTGTGTATTATTTGCCTGTTTTTTTCTTCTTCTGCTTTTCAATCACCTGATTACCTATCTTGTTTTTTCTCAATCTTCTTTTTTAAATGTGTTCTTTCTCTTTTTCTCCTCATAATATTTGGCACGTCTCCTAATCTCTTTCTCGACTTCTGTGGCGCCCAACATTCCTGCGCTTTGCTTCTCTCTCCTTTTACAGACAGCAGCAGGCAGACAGTGTTTGGTGAGTAGCTTCAGTGTGGCCAAAGAATGTGGCTTTTGGAGGTGCTCGCACTTTAAATCATTATGATGCTTAGTCATTTTGGGGAGACAAAGTCATGGGGAGGCAGGCTTGGAGGAAACAACTTCTGGCTGGAAACCGTGGAATAAATTGGATCGATTTGTTATATCCGCTGAGGACTAATCCTCAAATATGTGTGCTAAAAAATTTGCTTTTAATAGTAATACACATTTAGGTGTTTTGTTTGCATCCCTGTTTGGGTATTTATGTTATGCTATTCATTAACATGCAGAATATTTAGTTTAGAAGTGAAAGAGATGGTTTTAAATGGACAATAATTCTTCTTTCCTATCTGGCTTATTCCTTTATTTTCATTCTTTATTCTGCTTTTTTCATGGGAAAAGTTCTTTTTTTATTCAGTTGCTGCTTGATGACTCCTGCTTTTATCCCCATATTCATGAAAAAAATAGATGTTGTTGAAAATTCCAGATTTTTTTTTTCATTCTTTTCAAAACTTGAAAAGCTTGAAAGTTTAGGGTTTAAAGTTAAGCCTTAAAAGTAGTTTAAATGTGTTAAAGGTCGCTTTTTGAAACCAGAGTACACATTTTTTTATTTCTAAAGGCATTTTATTAGCTCTATATCAACTCAAAAAATTGAAAGCTGAAAATGCCAGAATTTTTGGTAACACTTTATAATAACTACACACTATGAATAATTTATTAAGCATTAGCATATAGTGAATTTGTTATTTGTTAAGCATTAACTCTACATTAATAAACAACTGCAGTCACAAATGCTGTATTCTCGATTTATAAACGTGTATAATTTGTTTAATAATAGTGTTTTCGTAATTTGTGAGTGATTTATTTTTCATTACTTAATTAAGTATTGCTTTATTTACAAACCAGTTGTATTTAGTAGTCGATGGTTTTTAAGATCATTCAGAATAAGTTAGTAAATGATTAATAAACTATTGAAATCAACGTTTATGCATCTTATTTTTCAGGCAATAGTATTGGTTACTATATATGTTAATAAATGCTTCAATAACTTATCTTTATGCAGTTTTGTGAACTAATCTAAAGCGAGGACTATTTATGCTTGGTAAATTCCTTATGAATGACAATTAAAGGCTCAGTATTAAATGAACAATAATCTTTGCAATCCTATCAAAAAAAATAAAATGACTGTAAAATGACTGTAAAAATTGGATAAAACAACAATAAAATAATTTAACAATATATTAGGATACAGCATTTCAATGTTATTTAGTGATGTTCAAACTTTACAGTCATTTTATTTTAGATAAGTTTGCAAAGATTTATTTTTGATTTGATACAGATTCATTTGTGTATCTTTAAATGAATAGAAATGAATAAAGTCATTTTTTTCCTGTATAATATATAACTGAGCCTTTAATTGTCATTTACAGAGCATTTATAAAGCATAAATAGTCCTCACTTTAGATTAGATCATTACCTAATTTTTTTTTTCTTATCAACTTTAAAGGCTTGGAGCGTTTACCCCTTTAACAGGCATCGTGACAACCTGGTTGACCTTTTATCTATAGTTAATTATTATTATACATTTTCATAATGTTTATCAGAGTTTTGTCAGTATATTATTCAATCCAAGATGTGGGCCAAAAAATAAATATAAATAAATAAAAAGAATAATTCAAAGTAGTGCACATTAAAGGGTTAAACATAATTAAAAATGTGTTCAAGTTAAGATTGTTTTTGAAACCAGAATGCACAGCTTTTATTTCTGAAGTCATTTTTTCTATATTCACTCAAAACAATGACTTATGCTACTTGTTCAAACCACTTATTTGAAATGAGTCAAAACAACACAATTCTTCATGTTTGGGGGACAACTTAATTGTTTCATGTTTAATCCTCTTAAATTTGTGAAAATGATTAAATTAGCTTATCGATTTGTGTTGGGACAACATGAAGGAATTGTGTTGACCCAAGCGTTTTGAAAGGGGTTTGTTTAGTGTAGCTAGTAGGATTGATATGAACTGTGTGTGTAAACTGGAGTGTTTCTCTTCAGTCCAGAGAGTGTGAATAGAGACTTGCCGGTTCTCAGGCATTATGCGTAAGTATACAGAAGCTCATTATGGTGACATGCTCTCTTTCTGCTTATACCAGCATGTGGCCGGGTGTTTGGGGTTTAATTCTGCGCTCAGAAACTGAGGCTGTGTGTGTTTGTGTGTGAACATTAACCTGTCATTTAACTTGAATTATTTTTCATTTCATGTAGTTCATAGGTCTCAATCTTGATTCCTGGAGGGCCGCAGCTCTGCACAGTTTTTTTTTTTATCTAACTCTAATAAAAACACAGCTGATCCATCTAATCAAGGTGTTCAAAACTACAAGAGACTATTATGCAGGTTTGAGTTGGAGGTGGTTGGAGATAAACTATGCAGAGCTGCGGTTCTCTAGGAGTTGAGTTCGAGACCATTGATATTGTGCAATTCTTGTAATATTAATGATTTATGTAAGGGCACATTTTTATGTTGCAGTAGCATGCATATTTTAACAACTATATAATTGTGTGCTTTGCTGCCTTGGTTATTGATATGTCAGTTTATTCTTTGCACACATGCCTCATTGTAACTCCTAAGTTTACTGAACTCGACTGTTGGATCACTATGGTGGAGAATTGAAGCTTGACTGAGTTAATCAAACTTGATCAGCTTGGTGGTTATCAAAACGAGAACTAATCATTCACGATTAATTTTATACTTTGATTTGTACTAGTTATATTTATGGCAGGGTGAATGGTGGGAAATAAAATGGTGGCTTGTGGTTTTGGGGTTCTAAATGGTGATGGTGGTGGTGGTGGTGTGCTTCGTCAGACACTGGATTGGATTTCTGTGGTGGGATTCAAGGGATGTCTAAATATGTAATTACTGTGTATTGTCATATTGCCTTTTTGTGTAAATGAACATTTTAATCTGTTCTATCTTTATATTATAACTTCTAATAGGTTAATTTTTAATGGTAGATTGATTTAAAACTGAGTCATAGTCTTTTGACTAAAATGGCGTATTTTATTTGTCTAATTATTCTTGCTTTAATGTATCTTATATATTTAGTATAGATTTAAGTTTATCCAAATCTAGTGAGATTATTTAAATTGCTCTGTCTTTAAGGCATGGTTCACTCAAAACTGAATATTTATTCTTCATTTACTCACACTCAAATGGTTTTAAACTTGTAGGAATTTCTGTCTTGGGGCGACACGGTGGTGCAGTGGGTAGCACCATACCGTGACAGCATGAAGGCCGCTGGTTCGAGCTCCGGCTGCGTCAATTGGCATTTCTGTTTGGAGTTTGCAAGTTCTCCCCGTGTTCACGTGGGTTTTTTCTGGTTCCTCCGGTTTCCCCCACAGTCCAAAGGCGTGCGGTATAGGTGAATTGGGTAAACTAAATTGTCCGTAGTGTATGTTTGTGAATGGAAGTGTATGAGTGTTTCCCAGTGTCTGTATTAGTGTTCCCATCTGCTGCATAAAACATATGCTGGATAAGATGGTGGTTCATTTGGCTGTGGCGACTCCAGATTAATAAAGGGACTAAGTCGAAAAGATAATGAATGAAGAAATGTCTGTCTTCTGTAGAATACAAAACAAGATATTCTAAAGAGTCTTTGCAAAAAGCAGCAAAGGAAATCTCTAGGAACTAAAAATACTATGGAAGTTGATAGCTAGTTTTTCCCAACATTCTTTATAATGTCATCCTTTGTGTTTAACAAAAGAAAAAAAACTCAAACCGTTTAAAGCATTTAAGTTTTTGGGTGAACTATGCCTTTAAGTTTATATTAATTAATCGATTCATAATTTAGGAAACAAAAAGATATTAGTGTTTAAACATACAATACAGAACTGTGAGATTTAACTTCTTTATCAAGAACATGTCAAGAACAAGAAACAAGAACATGTCACTTTAGCATAAGTAGGATCTGATGAATACATGAATACAATTTACCAGTACACATCTAATAATTTGTTTACTTGAAGGGAAAGTTCATCCAAAAATTCTGTCATCGTTTACTTGCCCTTGTTTTAAACATTTGGGTTTCTTTCTTCTGTTGAACACAAAAGAAGATACTTTGAAAAATGTTGGGAACCTGTAACCATTGACTACTATAGTAGTAAATAGTAAATTTACATTTTGAAGTGAACCATCCCTTTAAACCAAAATGGGTATTTTGACAGTTTTGTCTCATTCAAATCAAAGCTTGATAGCTCAGTTCCTAAAAAATCCTAATTGTCTTTATTGTATTGAATAACAGAGGAATCCGGAGAGACCAAAATTGCTCCAGAAATCCAGAAAGCTTAAATTTAGTACTGCCTTTGTGATTTATTGACCTACTTTACAGTATCCTGCACCCACTCATTTGAGAGCAGGTCTACAGTTTATAATGTATCTTCAGAATCAAACACTTTTCTTTATCACCCAAAACCCTCCATTTTCAGCTCAGTAAATGAGTGCTTCTTCTCCGGTGCTTTACAG
Seq C2 exon
AGACACGTTTTTGCGTTCTCTGGCTGTATCCGAGTGGGAGAAAACGAAGAAGTCGTCCGCCTTACAAGAGAAACCGAGTGAGAGTGAGGCACCCACTGAGACTGAGGAGCAGATCTCCCTCTCCACCACCACAACAG
VastDB Features
Vast-tools module Information
Secondary ID
ENSDARG00000051768:ENSDART00000073447:13
Average complexity
IR
Mappability confidence:
NA
Protein Impact
ORF disruption upon sequence inclusion
No structure available
Features
Disorder rate (Iupred):
C1=0.963 A=NA C2=0.714
Domain overlap (PFAM):
C1:
NO
A:
NA
C2:
NO
Main Inclusion Isoform:
NA
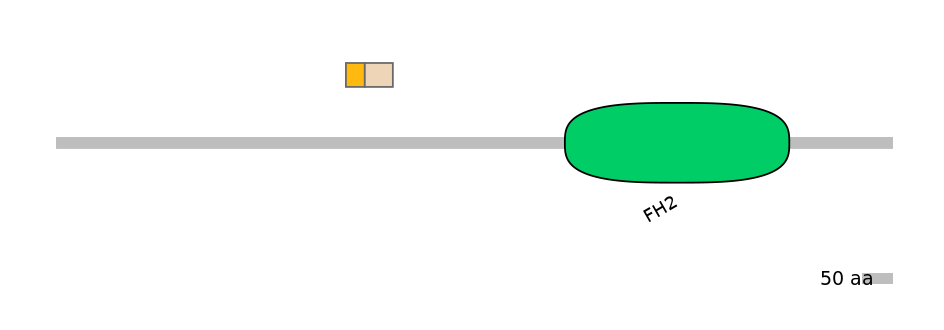
Other Inclusion Isoforms:
NA
Other Skipping Isoforms:
NA
Associated events
Conservation
Human
(hg38)
No conservation detected
Human
(hg19)
No conservation detected
Mouse
(mm10)
No conservation detected
Mouse
(mm9)
No conservation detected
Rat
(rn6)
No conservation detected
Cow
(bosTau6)
No conservation detected
Chicken
(galGal4)
No conservation detected
Chicken
(galGal3)
No conservation detected
Fruitfly
(dm6)
No conservation detected
Primers PCR
Suggestions for RT-PCR validation
F:
No suggested primer sequences
R:
No suggested primer sequences
Band lengths:
Functional annotations
There are 0 annotated functions for this event
GENOMIC CONTEXT[edit]
INCLUSION PATTERN[edit]