DreINT0133604 @ danRer10
Intron Retention
Gene
ENSDARG00000100594 | sez6l
Description
seizure related 6 homolog (mouse)-like [Source:ZFIN;Acc:ZDB-GENE-091204-162]
Coordinates
chr10:44816346-44818764:+
Coord C1 exon
chr10:44816346-44816540
Coord A exon
chr10:44816541-44818572
Coord C2 exon
chr10:44818573-44818764
Length
2032 bp
Sequences
Splice sites
5' ss Seq
AGAGTGAGT
5' ss Score
7.36
3' ss Seq
TTTCCCTCTCCTTTATTCAGTCA
3' ss Score
8.62
Exon sequences
Seq C1 exon
AGGTGTCCCGCAACGATTCGTGTCCAGATCTGCCTGAGATTCAGAACGGCTGGAAAAGCACGTCTCACGCAGCGCTGGTGCGCGGAGCCCACATCACATACCAGTGCGACCCCGGGTACGACCTAGTGGGCAGCGAAACACTCATCTGTCAAGTGGACCTGAGCTGGAGCACACAGCCTCCGTTCTGTGAGAAGA
Seq A exon
GTGAGTCTGGCAGACGTTTGTAAACTCACTACAGGTTCATTCAGATTCACTGCAGATTTTGTCAGATTCATTTCAGATTCAGTCGGATTCTGTGGCAGTTCAGTGCAGATTCATTGCACATTTAGCAGATTTTGCCAAATTCAGATTCAGACAAATTCATTTCAGATTCACTCAGATTCAGCGCCAGTTTAGTCAGATTCATTGCAGATTAAGCAGATTTTGTCTGATTCATTTAAGATTGAATCAGATTCTGTCAGATTTACTCAGATTCAGTGCCAATTTAGTCAGATTAGCTGCAGAGCAAGTCAGATTCAGCAGATTCAGTTAAATTAACTGTATATTCTGCTGTCTTACTTAACATCGAAGTGAATCAAAAATGTTTTTCAAAATTGATCTCGGACAAGAATGGGTGTTGCTCAGTAGTTTTAGGACAACTTTGATGAATGGTGATGATCCACTTCAAATGTTGACTATTGCAGTCAGATTCAGTGCAGATTCAGCCATGTTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCGGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCGGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCGGTCAGATTTACTAATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTCGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGATTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGCTTCAGTCAGATTTACTCATTTTGATTCAGATTAACTGCAGCTTCAGTCGGATTTACTCATTTTGATTCCGATTAACTGCAGATTCAGTCAGATTTGCTTTAGTTCGGTCAGATTCAGTGCCAGTTCACGCAGTTTATCTGCATTCAGATTTAGCTGATTTAGTCAGATTCAGCAGGTTGGTGCTGATTCATCCATGTTCAGTGCAGATTGATTCAGATTCACCCAGATTCAACAGATTTATCCAAATTCACTCGTTCAGTCAGATTCAGCAGAATGTCTCAGATTTACTCCGATTCAGCAGATGCATTCAATGCTCAGATTCAGTCAGATTAAGTCAGATTTGATTTGATGCATTCAAATTCACACATTCAGTCAGATTCAGCAAACAGATTCAGTCAGATTCAGTGCAGATTCACTCAGCTCTGTATCTTTTCCCTCTCCTTTATTCAG
Seq C2 exon
TCATGTATTGTACAGACCCGGGACATGTCGATCACTCCACACGCACACTCTCTGACCCCAAACTGCTAGTTGGCACCACCATCCAGTACAGCTGCACACCCGGATTCATCCTGCAGGGCGGCGCCACACTCACCTGCTATGGCCGAGAGCCCGGCACCCCGGTGTGGACATCCCACCTTCCACACTGCGTCT
VastDB Features
Vast-tools module Information
Secondary ID
ENSDARG00000100594:ENSDART00000172128:8
Average complexity
IR
Mappability confidence:
NA
Protein Impact
ORF disruption upon sequence inclusion
No structure available
Features
Disorder rate (Iupred):
C1=0.000 A=NA C2=0.000
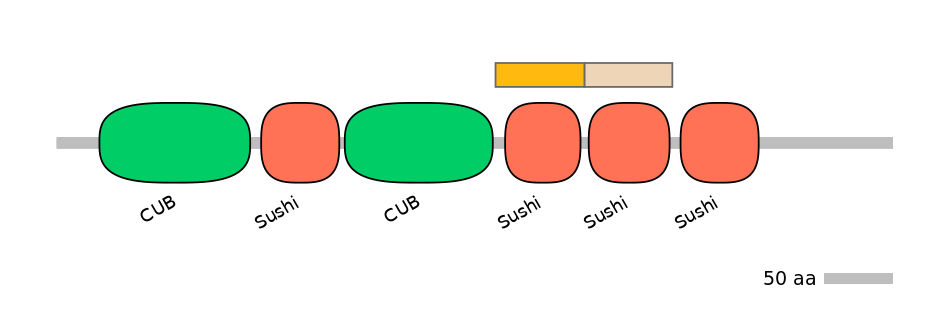
Domain overlap (PFAM):
C1:
PF0008415=Sushi=WD(100=84.8)
A:
NA
C2:
PF0008415=Sushi=WD(100=92.3)
Main Inclusion Isoform:
NA
Other Inclusion Isoforms:
NA
Associated events
Conservation
Fruitfly
(dm6)
No conservation detected
Primers PCR
Suggestions for RT-PCR validation
F:
No suggested primer sequences
R:
No suggested primer sequences
Band lengths:
Functional annotations
There are 0 annotated functions for this event
GENOMIC CONTEXT[edit]
INCLUSION PATTERN[edit]