HsaINT0049557 @ hg19
Intron Retention
Gene
ENSG00000187775 | DNAH17
Description
dynein, axonemal, heavy chain 17 [Source:HGNC Symbol;Acc:2946]
Coordinates
chr17:76472654-76475198:-
Coord C1 exon
chr17:76475107-76475198
Coord A exon
chr17:76472816-76475106
Coord C2 exon
chr17:76472654-76472815
Length
2291 bp
Sequences
Splice sites
5' ss Seq
CAGGTACTG
5' ss Score
9.04
3' ss Seq
TCATCCTGTGTTTGGGGCAGGGA
3' ss Score
7.27
Exon sequences
Seq C1 exon
CTTTGCATCAGAAAATCACGGCAACATTTCTTCCCACGGCCATTAAGTTTCATTATGTCTTCAACCTCAGGGACCTCTCCAATATTTTCCAG
Seq A exon
GTACTGCTCTTGTAGCTTTATGTCATGCCTGCCTTACGGTCCAGTGCCTGATCCACAGGTGCTTTCCTCATCACAGCTCTCAGGGAGGTGTGGACTGATGATGGTGGTGATGGTGGTAATGATAATGATGATAATAATGGTGGTGATGGTAATGGTGGTGGTGACAGTGGTAGTGATGGTGGTAATGGTGATGGTCGTGATGATGGTGATGTGATGATGGTGGTAGTGATGGTTATGATGATAGTGATGGTGATGTGATGATGATAGTGATGGTGGTGATGGTGATGATGGTGGTGATGATGGCAATAGTGGTGGTGATGGTAATGATGGTAATAATTGTGATGATGGTGGTAGTGATGGTAGTGATGGTGGTGATGGTAATGGTGATAATGATTATAATAATGGGGGTGATGACGGTGGTGGTGACAGTGGTAGTGATGGTGGTAATGGTGATGGTCATGATGATGGTGATTGTCATGATGGTGGTGATGATAGTGATGGTGGTGGTGAGGGTGATGGTGGTGATAATGATGGTGATAGTGATGGTGGTGATGATGGCAATAGTGATGGTGATGGTAATGTTGGTAATGATGGTGGTGATGATGGTGATAGTGGTAGTGATGGTGGTGCTGATGATAATAGTGGTGGCAGTGATGGTGGTGGTGATGGTGATGTGGGTGATGTGATGGTGATAATGGTGGTGGTGGTGATGTGATTGTGGTGATGGTGGTGGAGATGATAATGGTGATGGTGGTGGAGATGATAATGGTGATGGTGGTGATGATGGTGGTGATGGTGATGTGGTGATGATGGTGGTGGTGGTGATATGATGGTGATAATGGTGATGATGGTGATGTTATGGTGGTAATGGTGATGATGGTGATGTGATGATGCCACGTGGTGGTGATGGTGGAGATGATGATGATGGTGGTGGTGATGGTGATGATGGTGGTGGTGATGGTGGAGATGATAATGGTAATGATGGTGGTGTGATGGTGGTGGTGATGGTGATGATGGTGATGATGGTGGTGGTGATGGTGGTGATGATGATGGTGGTGGTGATGGTGGAGATGATAATGGTGATGGTGGTGATGATGGTGGTGATGGTGATGTGGTAATGATGATGGTGGTGATGATAGCGATGGTGGTGGTGATGGTAATAATATTTCCTCTCATTTACTGACTGCTTACTCTGAGTCAGAGATTGGGTTTGGCCCCTTACATTCCCATTTTCTCCTTCTCCAACAACACATAAGTTTGGTGCTTATATCCCCATTTTACTGAGCAGGAGTTTGTGGCTGAGGGAGGCTAAGTTATGCCCACCTCCTCCCACGTAAAGTATCAGCCCAGCCGGGTGCAGTGGTTCATGCCTGTAATCCCAGCACTTTGGGAGGCCAAGGCGGGCAGATCACGAGGTCAGGAGTTCGAGACCAGCCTGGCCAACATGGTGAAACCCTGTGTCTACTAAAAATACAAAAATTAGCTGGGCATGGTGGAGTGTGCCTCTGTAGTCCCAGCTACTCGGGAGGCTGATGTGAGAATCACTTGAACCCAGGAGGCAGAGGTTGCAGTAAGCTGAGATCGTGCCACTGCACTCCAGCCTGGGCAACAGAGTGAGACTACATATCAAAAAGTATCAATAAAAAAAAAAATAAAGTATCAGCCCAAATGCCATGCTCTCACAGAGGCCACTCCTCAGCCCTCACACTCTGTCTTACTGCCCTGGGTTTGTCATTCAGAGCATACACCATAGTGTGCAATTACCTTATTTCTTTGTGTACCTGGCCACCTCCCCCGCTAGCATGTCAACTCCACAGCGTCAGGTTCTGTTGCATCATCGCTGTGCCTGGGAAATTCCAGGCCCTCAATAAGCTCTTGTCGAGCTTGCCTAGAGTCACTTGCTTAGTAAATGGCCAAGCCAGGACTGGACCAGATAGGCTTGCTGTGGTACCCAAGCTTGTGACCTGCCCTATGGCATCGGTCACCTGCTTTCCTTTGCAATTAGCCTCCCTTGTCCCTCCTCCACCATTCACTCTTCTGGAGGATGAGGCCCCAACCCTGTCCTATGAGGAAAACTCCACATCTCTCAGGACACACACACACACTGGCATCCGGGATTTTTGGGACCTCCGTGATGAGTGGCTTCCCTGGCCCCAAGTGAATGCTGTGCCAACAGCTGAACAGGTCCCCTCGCCCCGGGGGAGGGGGCTGCTCCTCAGGGTCATCTGTGTCCAGCTCTGGGCTCGCCTTCGTAACAGCTGACGGTGCTGATCATCCTGTGTTTGGGGCAG
Seq C2 exon
GGACTCTTATTTTCCACAGCAGAAGTTCTGAAAACCCCACTGGACCTCGTCCGCCTTTGGCTACATGAGACTGAACGAGTGTATGGTGACAAAATGGTTGACGAAAAAGACCAGGAAACATTGCATAGAGTCACCATGGCCTCCACCAAGAAGTTCTTTGAT
VastDB Features
Vast-tools module Information
Secondary ID
ENSG00000187775-DNAH17:NM_173628:51
Average complexity
IR-S
Mappability confidence:
NA
Protein Impact
ORF disruption upon sequence inclusion
No structure available
Features
Disorder rate (Iupred):
C1=0.000 A=NA C2=0.000
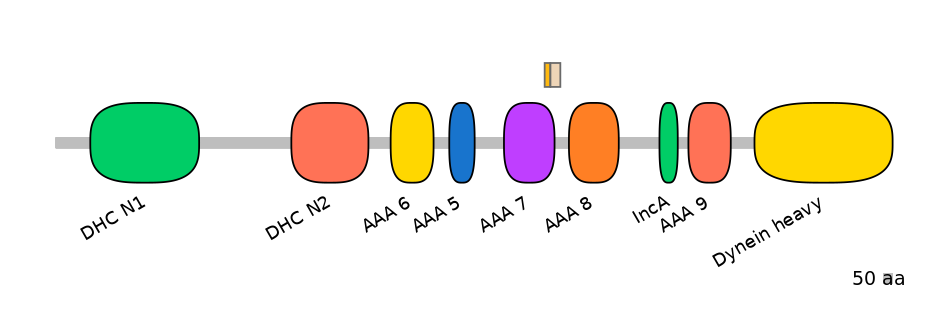
Domain overlap (PFAM):
C1:
PF127752=AAA_7=FE(11.0=100)
A:
NA
C2:
PF127752=AAA_7=PD(8.1=40.7)
Main Inclusion Isoform:
NA
Other Inclusion Isoforms:
NA
Associated events
Other assemblies
Conservation
Rat
(rn6)
No conservation detected
Chicken
(galGal3)
No conservation detected
Fruitfly
(dm6)
No conservation detected
Primers PCR
Suggestions for RT-PCR validation
F:
No suggested primer sequences
R:
No suggested primer sequences
Band lengths:
Functional annotations
There are 0 annotated functions for this event
GENOMIC CONTEXT[edit]
INCLUSION PATTERN[edit]
SPECIAL DATASETS
- Autistic and control brains
- Pre-implantation embryo development
Other AS DBs:
FasterDB (Includes CLIP-seq data)
AS-ALPS (AS-induced ALteration of Protein Structure, links to PINs)
APPRIS (Selection of principal isoform)
DEU primates (Only for human)