RnoINT0060947 @ rn6
Intron Retention
Gene
ENSRNOG00000012881 | Fgl2
Description
fibrinogen-like 2 [Source:RGD Symbol;Acc:620170]
Coordinates
chr4:10323607-10329241:-
Coord C1 exon
chr4:10328636-10329241
Coord A exon
chr4:10326687-10328635
Coord C2 exon
chr4:10323607-10326686
Length
1949 bp
Sequences
Splice sites
5' ss Seq
CAGGTAGGT
5' ss Score
10.28
3' ss Seq
TTTCCTTCCTTCCCTTTCAGTTC
3' ss Score
13.39
Exon sequences
Seq C1 exon
GGCTGGGCTGCTGCACTGCAAGGATGAGGCTTCCTGGCTGGTTGTGGCTGAGCTCCGCGGTCCTCGCTGCCTGCCGAGCGGCGGAGAAGCACAACCTGACTGAAGGGCTGGAGGATGCGAGCGCCCAGGCTGCCTGCCCCGCGAGGCTGGAGGGCAGCGGGAGGTGCGAGGGCAGCCAGTGCCCCTTCCAGCTCACCCTGCCCACGCTGACCCTCCAGCTCCCGCAGCAGTTCGGCAGCATGGAGGAGGTGCTCAAAGAAGTGCGGACCCTCCAGGAAGCTGTGGACAGTCTGAAGAAATCCTGCCAGGACTGCAAGTTGCAGGCTGATGAACATCCAGACCCTGGCGGGAATGGAGCAGAGACGGCCGAGGACAATCGAGTCCAGGAACTGGAGAGTCAGGTGAACAAGCTGTCCTCAGAGCTGAAGAATGCAAAGGAGGAGATCCAGGGGCTGCAGGGGCGCCTGGAGTCGCTCCAACTGGTAAATATGAACAACATCGAGAACTACGTGGACAACAAAGTGGCAAATCTAACCTCTGTGGTCAACAGTTTGGATAGCAAGTGTTTCAAGTGCCCCAGCCAAGAACACAACCAGCCAAATCCAG
Seq A exon
GTAGGTGCAATGAGGTTCTTCGAGTTTGTTCACGAAAGCTGTATAGCCAGATAGTGGCCGTAAACATTAACCCGAGGGACTATAAGTTAGTCAGACTTTCACCTGTTAAGTTATGGCAGAAGCAACAAGTGTTTTCTCAAATGAGACAGCAGAAATGGTAAGTGATCTAATGACAAAAATGCCATTAGTTGTTCTCCTTAGAGACCACTTGCAGTCTCTAGACCTCCCTGTTAGGCTGACTTGCAGACGAGCAGAAGCAGATTCCTCCCAGTTTATTGCATTTGAACACAGGATGGATACGGCAAGGTTCCTGGCTATGCTCAAGCAGGTCCCTGGAAATCCGGTCTTAGGTATCTGATGGCTTCTTCTTAGATCAGTTCCTGAGCTTTATAAGGCTTTCACAAACAGTCTGCTGGGCTAGACTCTCAAATACCTAGTGAGAATGGGAGAGTTTATACATGGAATAAAAGCATCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTGTACAGAACTTTTCAGTTTGAGACTTACTGGCACATCCATCCTCACCATCCCAGTTAGTGAATGTTACCACTATTTAAGATCACAGACCTAACTGCCTTCTGTGCCCATGACATTGGTTTCCACGATTCCTGGATTCTGAGGACCTTTGTGGGTGGGTTGCCACACCCTGAAAGGTCACCTCTGAGTTATTATCAGTGCTTATCCTGTGTAGGGACATAGTAGAGGTCACATTCATTTGGAATCTCCTGGAGACGGTAGATCACCATGTCATAGAATGATGTCACTAATGACCAAGGTACACATTCATGTTAAAGAGACATCACAAGGCGTATCTTAAATATCACATTGAACATTACTTGTAATGACACAAAAAAATTCTATTATTTGCTTTTGTCCTAAAAGTTTAGGACTCTTCAGAGCTCTACATACATGCAAACAGATTTCTGTGTCTCACGGGTTATCTTATATTAAACTTTCTTACCTGACTCCAAGTTTTATTAAAATTAACTGGAAACAAATAGTCGGTCTCTAATCTCTATAAAACCACCAAATGGCTACGCCGAGCATAATTATAATTACCTGCTGCTGCTTCTCCTAGAAACTAAACTGTGGGATACTGGCTGCCCATTCCTTCCTGAAGAGTAAATTCAGGATGACACTGCTTTAGTATTGGGGAGCACACACACCCCTAAGTAGAGTAGCTTTATATTGTTCTCAGATGAAATGAGAGTGAGTCTCAGAACAATGCTGTGCGCTCAGGCTGAGGCTTCCTTTGGAACATATGTAACGATTTCTGTAACAATTGCTTTAGCGATGGCTGACTGTGTCCTAAAAGACTTGGACTCACCTCAACTTTAAGACCTAGGAATATAATGATTATGAGAGTAAAGCTCTCTGTCAGGCAGATCTGACTTTGTGTCCCAGATCTGTCACTCAACACTTAGTTGCTGTGGGAAAAGTCTCTGATCTTCTAGGAGACTGACTGACTAGATAAGCAAGCTGACCATAATACCCGCAATACCTCACCAGAAGGTTGATGTGTGCGTTCAATGAAAACTGTTACAGTGTTCCGCAGAGTTTCTGGAGGTCATTGGTCCTCATTAAACCTTGCATACATTTACTCTTACTACTGTTTGCTGTCAGTGTTGCCACCATGGTTGCCATCCAAGGCAGACAGCCCTGTGTACTTGATGGTGCCGGTTGTTTCTGAGGCCTTAGTTAGCGTCTGTTAACCAGATTCAGGATCGTGTATCAGAGCCAGGTTCCATTATACCCAGCAAGTAAATTGTCCAGTTGCTGTTGAGAATCCTTGATTCTCAGACTCATAGCTTGATTAACACATAGCTCTGATTAACGAAACCCCATTTTTTTTAATGCTTTCCTTCCTTCCCTTTCAG
Seq C2 exon
TTCAACATCTAATATACAAAGATTGCTCTGACTACTACGTGTTAGGGAAGAGAAGCAGTGGGACCTACAGAGTTACGCCTGATCACAGGAACAGCAGCTTTGAGGTCTACTGCGACATGGAGACCACGGGTGGAGGCTGGACAGTGCTGCAGGCTCGCCTTGATGGGAGCACCAACTTCACCAGAGGGTGGAAGGACTACAAAGCCGGCTTCGGGAACCTTGAACGAGAATTTTGGCTGGGCAACGATAAAATTCATCTTCTGACCAAGAGTAAGGAAATGATTTTGAGAATAGATCTCGAAGACTTTAATGGTCTCACACTTTACGCCGTGTACGATCAGTTTTATGTGGCTAACGAGTTTCTCAAATACCGGTTACACCTCGGTAACTATAATGGCACGGCAGGGGATGCCTTGCGTTTTAGCCGACATTACAACCACGACCTGAGGTTTTTCACCACCCCAGACAGAGACAATGATCGGTACCCCTCTGGGAACTGTGGGCTCTATTACAGCTCAGGCTGGTGGTTTGATGCATGTCTCTCTGCCAACTTAAATGGCAAATATTATCACCAGAGATACAAAGGGGTCCGTAATGGGATTTTCTGGGGCACCTGGCCTGGTGTAAGTCAGGCACACCCAGGGGGCTACAAGTTCTCCTTCAAGAAGGCCAAGATGATGATAAGACCCAAGAGTTTTAAGCCATAAATTCATGCTCGTTTCTCTGGGCACTCACTATCTAAGAGGACAGTGCATTCCTCCAGCCTTGACTGTATGCCTCAGTTTATATCCCCTTCCTGTGGCTAAACACTCCCTTTTAAGCTTTACAGCTTTAAAATAAAGCTGGAAATGTCTAAAACAGACTCTTTGGTGGCTGTTATATGAGGAATGCTCGAAAGCACTGGAAATAGTGACGATTATACATTATAATTGCAAGACTTTTCATTTTTATTAGTTGAAAAGTTTCCTACTATTTTTATTATTTTTTATAATAAAAAACTAAATTATTCAGCAAGCTAAATTCTGTATACTCAAGTTTTATTTTCACTAGGGCTAAACGCACACATTTGAAAATACACCAGTCTTTTTCAGGGTACAGACGCTCGTGTATCTGGAGCTAAGAACTATAGTATTTTCTTTGGTCCAAGAAATAGTTTAGTTCTCAAAAAGAAAGTCAGGGATGGGGTTTTGGTGGGCAGGGATTAAATAGCTAAATTTATTTATCTAATGCAAAATATGGCATTAAAATGCCTTAAAATACAGTAGCATAATATATGTCTTTTTCCTCTCCAAAATTGGAAAATACTGCTACCCTGGAGATTGTGGTTTCGTGGTGACCCACGTACTGTTTATAGCCTGTTAGACCCTGATACAGAAGCTGCTTTGTCATCTCCTCTCTGTGCACGATAGTGTTTAGTGTAAGGTGCGAGACTACTGTAAGGCTTCCTTGGAGAAAGGCATGGTAAGGGAAAACAGCGTTTAATTATACTTTGAAGGCCAATGCTAGACCCAAAGCAAGACTTGAGTTGTTGAGGAATCAACTTCCTAGGAAGCAGACTTTTCTAGAACAAATATATTTATTGTGATACATTTTGATAGAGTAGTTCAAATATTATTCATTTTAATCCTAAGTCGTCAACTGCTTATTTCATTCTCAGAAATTCACAGAAAAACTATATATAATTTTAGTCCAGTGTAAGTGCCTGGAGTTCCTTGATTGTGGAATGCTGAGGGCCATGTTAAAAGGCTTGGGAGGAAGTTTCATTTATCACTGTAGAAAAGGATCACTTCAGAGAGGAAAGATGCTCTTCAGTGGAATCTGAGGAACTGTACGCGGTCAGTCATAGGAGGAGGTGATCAGACAGGGCGCAGTGTTTAAGCAAAAACTTTGATCTTTAAGTTTTGGTAGATTTCTAATATACTAACCAAGCCTTTGAAATATAAGCACAAAGATTCTATTTTAGTTAGTCTTTAAATATAACTAGGAAGGACAGTTATTGACTTGACTACTTAAATTTTCAAATACGTAGCCGCTGTAAAAAAAAAAAACTTTAAAAAGAGCAAGATATTATTTTAAAAATATGCAACATGAAGTTTTCAGCTGAATACAATGACAATTAAAGTATTAATAACATAATTAAATTACCAGAGTAAGGTGAATGCACGCTGAATTAAATGACCAAAATATAAAATATAGATTTCCAAGACTGTTTACCTGCTTGGCAAAATATTGAATGTAAGTAGCAATAATTAGTTTTATTTCTCCTTTGCTTTTTAAAAAAGAAAAAGAAGATTTTGATATTATACAATTGATGTGTTTCAAATTCTGTTTGTTATATGTAACCATAGAAGTGTAATGACATTTCCGTCATGAAAATGGATTTATTATAGCAAAACCCCTATGCATTCCTCTTTAGTGTGTCTTTTTCAGTGTAACAATGAAGCCATGTGAATGCTCTATCTTTGCACGTCCAATGGATTCAAATGATGTTTTCTATTATACTCTAGAGATTTTTCTAATGAACTCTTTAACACAGAAATTTATTTTTGGAATCCCTTTCTGTCACTTATTTAAGAATATATTGTAGGCTTTGATAACCTTTAACCTGTTTGTTGGAATAGTCCTTGGCCTTAACTATTCACTAATGTATGCCAGTCCTCATAGGTGGACACTATTGGCAAGTCTGTGTTTTAGAGTCTTGGCTTCATCTCGGTGGCTAACGCTGGTCTTAACTATCGCTTTTTGGCTGAACATTCTTATCCTTTAAAATGCTGAGTGGAACTAATTTCTTAGGAATCTGGTTGAGTCACCCATCACCGAATGAGAAAGCCATTTTTCTGAAAAGTAATAATGCAGCAAATGTCTTCAGCCTCTGATGGACAGAACTTGTGTACAAAGCACAGGATGCATGGGAGTCACACACAGTGCTGGCTGGTCCGGGGCTCTTAATAGCTGCCCCTCGCCCCTCAGAAAGTTTTCAAGCTGGAATGGATATGTGTGATTACAATAAACTCTCGTTTAGGATTTCTTTTCATTGATGTATAATTAATGTTACATTTCATGTCTCATTAAAGCCTTGT
VastDB Features
Vast-tools module Information
Secondary ID
ENSRNOG00000012881:ENSRNOT00000017232:1
Average complexity
IR
Mappability confidence:
NA
Protein Impact
Alternative protein isoforms
No structure available
Features
Disorder rate (Iupred):
C1=0.226 A=NA C2=0.004
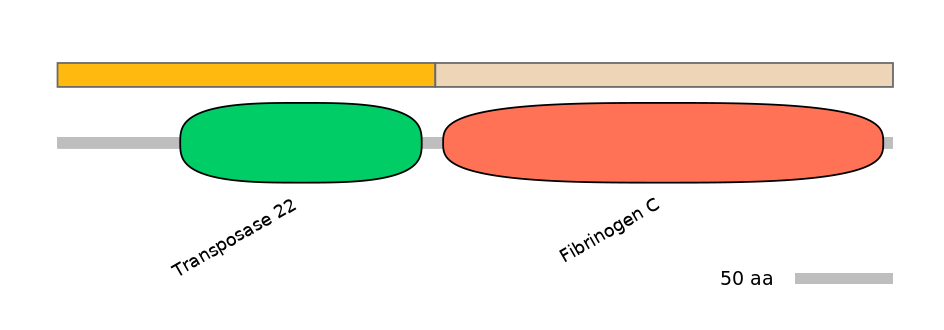
Domain overlap (PFAM):
C1:
PF131661=AAA_13=PU(98.0=74.9),PF097304=BicD=PU(95.3=73.3),PF039618=DUF342=WD(100=63.1),PF029949=Transposase_22=WD(100=64.1),PF0080420=Syntaxin=WD(100=60.0),PF060097=Laminin_II=PU(94.4=43.6),PF053776=FlaC_arch=WD(100=17.9)
A:
NA
C2:
PF131661=AAA_13=PD(1.3=0.8),PF097304=BicD=PD(4.0=2.5),PF060097=Laminin_II=PD(4.4=1.7),PF0014713=Fibrinogen_C=WD(100=96.2)
Main Inclusion Isoform:
NA
Other Inclusion Isoforms:
NA
Other Skipping Isoforms:
NA
Associated events
Conservation
Chicken
(galGal3)
No conservation detected
Fruitfly
(dm6)
No conservation detected
Primers PCR
Suggestions for RT-PCR validation
F:
ACGTGGACAACAAAGTGGCAA
R:
TGCCCAGCCAAAATTCTCGTT
Band lengths:
342-2291
Functional annotations
There are 0 annotated functions for this event
GENOMIC CONTEXT[edit]
INCLUSION PATTERN[edit]