DreINT0078467 @ danRer10
Intron Retention
Gene
ENSDARG00000062049 | hmha1b
Description
histocompatibility (minor) HA-1 b [Source:ZFIN;Acc:ZDB-GENE-071213-2]
Coordinates
chr22:17601742-17603336:-
Coord C1 exon
chr22:17603226-17603336
Coord A exon
chr22:17601823-17603225
Coord C2 exon
chr22:17601742-17601822
Length
1403 bp
Sequences
Splice sites
5' ss Seq
ATGGTACAG
5' ss Score
3.5
3' ss Seq
TTTTTTATGTTTGTCCCTAGCCT
3' ss Score
10
Exon sequences
Seq C1 exon
CCACACATGCCCTTCTTCACCATCTACTCTCAGGCACTGGAGCAGGATCTGGAGCAGAGCAATGGCATGCATCAGGCTGCCGCAACTCTACAGAACCAGACCTTTATCATG
Seq A exon
GTACAGAGACAAAATGACCACTGATTGCCCTGTCAATAGAAACTACACCAATTAGCATTAGTTACTATGATGTTCATGATTAATATAATTTAATGTTCATGATTAGTTAATATCATACTATTAATAGCATTTTTAGTTGTTGTTGTTACATTACTAACTTATGTAATACAGTGGTATTTGCAGTAACGATTAACATGAAAATTAACAATATACATAATAATACAAAATAAGTAGTAACTATTATACAAAATATATTAGTTTATTAACCTATTAATATGAAATATACAATTTATAATAATAATAATAAAAAAAATATGTATATTAAAATTAAATGTTCTACATGAAAATAAAGTCTGTATGATATAATTTGATTAAATGTTAATTCATCATTATCATCATCATCATTATTATTATTATTCATAATACAATTTTATAATAATAATAATAATTATAATAATAATAATTATTATTATTATTATTATTATTATCATTATTATTGTTATTGTTATTATTATTATTATTAATATTATTATTATTATATCCATAATAAAATAGTATTGTTAATTTTAATATTATTACTCATCATCATCATCATCATTATTATTATTATTCATAATATAATTTTATAATAATAATAATTATTATTATTATCATTATTATTATTATTATTATTATTGTTATTATTGTTATTATTATTATATCCATAATATAATAGTATTGTTAATTTTAATATTATTACTAAAATTGTATATTTATTATTATTATTATTATTATTATTATTATTATTAATAATAATAATATCACACATAATAATAATTATTATGATTATATTCATAATACAATTTTTAAATATTATTGTTAATAATAGTGTTCATATTATTACTATTATTATTTTATTAATTTTATATTTATTATATTATCATTATTATCATTATTAACGTTTTATATTATTTTTATTTTTATATTTTATTATTATTATTATTATTATTATTATTATTATTATTAAAAATAATAAATTTTATAATCATAATTATTATTTTTATTATTATTATTATTATTACAATTGTTTATTATTATTGTTTTTTATTATTATTAATAGTAACAATATTTATTATTATTGTTGTTGTTGTTGTTAATAATATAGTTTATCAGAATTGTGCTTTTTATTGTTAAACAATATTAACCAATAATAAAATTATTTTTAATATAATTTTAAATTTTCTTTTAATATTTTATGTATATTATTTTTGTAGCTGAATATTCTGTATCACTTGAATTAAGATATGCAACTTAAAATTGCTGTGTCTGCATCCCTTTCTGTTTCATTTTTTAAAGTTCTACTTATTATTATTATTATTTTTTTTTTTATTGTTTTCACTTAAGTTTTTTATGTTTGTCCCTAG
Seq C2 exon
CCTCTGATGGTCCGCAAACAAGAGCATGAGAAGAAACGCAAAGAGATCAAGGAGCAGTGGCTGAAGGCCAAAAGAAAACTG
VastDB Features
Vast-tools module Information
Secondary ID
ENSDARG00000062049:ENSDART00000132565:3
Average complexity
IR
Mappability confidence:
NA
Protein Impact
ORF disruption upon sequence inclusion
No structure available
Features
Disorder rate (Iupred):
C1=0.216 A=NA C2=0.185
Domain overlap (PFAM):
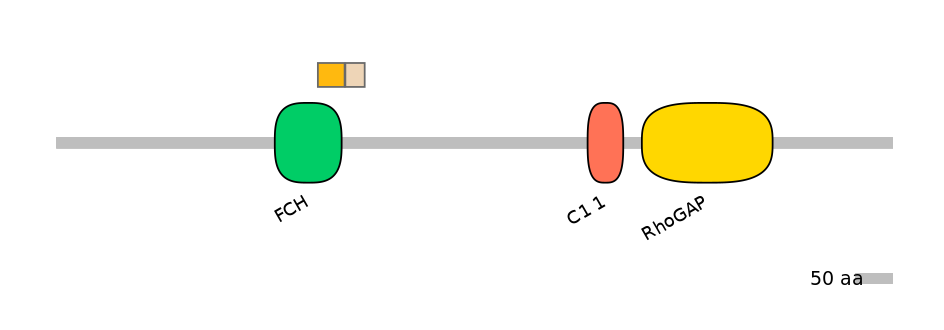
C1:
PF0061118=FCH=PD(35.2=86.5)
A:
NA
C2:
NO
Main Inclusion Isoform:
NA
Other Inclusion Isoforms:
NA
Associated events
Conservation
Fruitfly
(dm6)
No conservation detected
Primers PCR
Suggestions for RT-PCR validation
F:
CACACATGCCCTTCTTCACCA
R:
CAGTTTTCTTTTGGCCTTCAGC
Band lengths:
191-1594
Functional annotations
There are 0 annotated functions for this event
GENOMIC CONTEXT[edit]
INCLUSION PATTERN[edit]