DreINT0037266 @ danRer10
Intron Retention
Gene
ENSDARG00000079286 | bcr
Description
breakpoint cluster region [Source:ZFIN;Acc:ZDB-GENE-040724-43]
Coordinates
chr21:17152802-17156388:+
Coord C1 exon
chr21:17152802-17152854
Coord A exon
chr21:17152855-17156247
Coord C2 exon
chr21:17156248-17156388
Length
3393 bp
Sequences
Splice sites
5' ss Seq
CATGTGAGT
5' ss Score
7.83
3' ss Seq
TTTCTATTGATTGTCTACAGGAT
3' ss Score
9.41
Exon sequences
Seq C1 exon
CTCTGCTCTATAAACCAGTGGACAGAGTGACCCGGAGCACGCTTGTTTTGCAT
Seq A exon
GTGAGTCAACCTTAGTTTATTATAATATATTCTTTAGGCATTACTTATCATTTTTACTATTACTCATGTTGTACACTCCAAATGCCAGTGCCTTGTGAAGTTGATTTCCCAAGGTATTGGGCCTGTTTAAATTAGATTTCATATATAGTATAGTGTATATTTTAGGTTGGTCTATGGGCTAATGTATAATTATATCCATGTATTAATCACTTTCCCATTGCCAACATGTAAACAGCTTTGGCATGAATTTTTGATTTTTATATGGAGTGTTTTATGCAGAAAATTAAACTGATGTGACGTTTACACCAGGGTGCAAATTACAGGGGGGTTCGGGGGGGATTGACCCTCCTGATTAGCACTTGATCCCCCATAAAGGAAGTCAAAACAAGATGTATGGGGGGTCAGTTCTTTAATACTGAGAAATATTACACTTTGATCTGTATTTCAAATATTATTTTTAGTAATTAAAATATTATATAATACAAATATATATTTGACCACCCCTATAAAAGTTCATACCATTGTTAATGCGTAATTGCGCCAATCAGTCTATTCGAGGAAAAAAAGTCTGACAGCAGCAGATCTAGAGAATCAATAGACATTGTGTTCACAACACTTTGTTTTCTTGTAAAATAGGTGTTTGTATTTATATTTAGCCTGGAAATCAAATCAAATTAGACATATAGTTTGTAGTTTAACCTATAGTAACATCTGAAATAGCTAATCGGAGAATACTGTAGGTGAGAATACACTAAATATCCCTCTAAACACTGAAAACGTATATGCATTTTATTAATAAGCTAGATGTAATTAAAAATAAATACATAAATGACTGAAGCATGTGTTACACAACCCGAAACAGTTGACCCCCTGATCGTCAATGTATAATTCTCACAGTGGTTTACGTCCCAAATTGCCTTCCCCACTTTTCTGTAACAATAAAATGAATGTTTATACAGTAAGTAAGATTATGCTAAAAGAATAATCCAAAGAAAAGTGATTAATTCAAACTGTGGTCAGAACTATGTAATCGATATTCTCAAATATACTTTGATTTTAAACTACATCTATATTTTAAAATCATAACAGTGTCATTTGAATTACCTCATCTGTTGATAATATGATGCTAATTAATTGGAGAGCTTGAGCAAACAAGTCCACACGACTATGATCAGATTTTTTTTCCCTAAATACTGGCATTTTTGTGGTGTTTACTTTGTTATTTAGAAGAAATCATATAACACATATTTATATATTTATAAATCTAAATGACACTCTTAGCCATGTGGTCTGTGCCTGGATGTGTATCATCGGTATATTAGAACGACATTTTAAAAATCACACATGTACTGTAATTAAACAATGAATACAACTTTGCAGTAAGTATATCAGGGCATTGAATCACACAATGTCACACTCAAGTGGTTCCAAAGTGGTTTATGAATTTCTCATGAACTCAAAACAAGATATTCTGAAGAACGCTGATTTTAAAAAAAGCCATCGACTCTAACAGCCCTTGAGGAAAAAACAGATAAACTCAAAAAATGACTTTTGCTGCATGTTCTACTGAATTATTCAAAATTAATATTTGATTTTCTGTGGAAACAACACAACTCTTAAGAGTTTTTTGGGAAAACTTAATTTTTTATGTTCTTTCCACCTTAATTTGTAAAAATGATTAAGTTAACCTAATCGATTTGTGTTGTGACATCATGAAAGAATGGACCCTTCACTAAGCCCCGCCCTCCTTAGTTACTGTTGTTACACCTATCAAGCATTCGTGCCTGACACGTCTATTACACTATGTATGAGCCAATGTCAGACATTCCCAGAGATATAATTAATCATTTTTGGTTAACAACTGAGATTTATGAGAAAGCCAGAGAAAAAAGGACTGCAGTATGCATCACAGGGGTATATTCACGACTAAATACGCAACAAATTCGAAAATAATTTAATCATAGTTGAAGCTACAGTAGGTTAGCCTAGCCGCCTCATACACTGACCATTCAACAGCTTAGTTCATGCACAGCAGGATAGGTGACAATTAAAAATAATCTTATAATAACAAATTAAACTGTGATTTCGTGATTTATTTAACATAATTCATTCATAGAAAGAGCAGCCAGAAAGGGGAAATTGATGGATTTGTGCCAAATATTGGCACCATTGACCCATAACAATGTACAGCACCGATAACTGTCAGTATGTTTGTGTGCTTTTTATACCATTATTATTTTAATTTGCAATTAATTGGAAAAAATAAATAAATTGAAATGCAAATCAAAGTATAAACAGCAGAAGTGAACAAATAGATTTTCCCTACCAGCAAATGTTAATCCGACACCAAATATAAAAGCAGGCACTAACCCGGTCTAACGCCGTCACCAATGGCTAAATCTCCAAAAGTCAAACGAAAATAATGGTGATTTGTAGCTCTGAAATGGACATGAGGGATATAAATGACTAAAAAAACGCTGCGGGTAAAGTATATAAGTGCTCAGTTTGTGATCACATCTCGGTTTTGTTTATATGTAATTTGAAACATTCATAAATTAAAACAGATGGAAATATGACTGGATTGTGCTCGCTGTTATTGCTATTGCAAAGGCATATGCACAATGCATTGTTTCCCTCGGTCGAAAGCTTGACAGACCTCCTGCGAGTAGCTATGGTTTTCAGGTGTGGCTTAGCGAAGGGTCAATAGTGTGGAACCCAGCATTTTTTACAGTGTACCATGGGAGGTAATTGCTGTTCGCAATATTATTGAGTAAGTTTATCCGCATTTGTGTTTAACAAGAAAAAAAGAAACTCTAAACAGGTTTGGAACAAATGGAGGATGAGTCAATGAATTAAAATAGAATTAAATCTTTGGATGGTTATGCTCAAGTACAATCACAAGCATTTCTTGCTGCAATTCACTTCATGGTTTCTGAATTCTGTAGCATTTAAGCCAAGAATTCAAAGCTTTTAAGTACAACTCCTATGTCGTAATGAATTAACCTACTGGTAACTGGTTCTTTTATTTAGAGGGCAGTAACTAATGCACCTTACACTGTAAAAAAACAATGGGTTCCGCACTATTGATTTTTGTTGGAACAATGTGAAAGAATTAAGTTAATACATTGGTTTTTACAAACTGAAGTGGATTGAACATACAACAGTTAAGTTGACCCCAAGAAAACTTAAGAATTGTGTTGTTTCAGCTGATTTTAAATAAGTAGTTTGAACAAACAGCAAGCATAAGTTTTTTGAGTGTAGATCAATGATATTGCAGTATTTCCTATTCATAGTAATTCACCGTCTGGGTTCACCATCTTGTACAGTATTTTCTTTTGCATGATCGAAGAAAAAAATGTACTATCTCTTTGATTTTCTATTGATTGTCTACAG
Seq C2 exon
GATCTTCTAAAACACACCCCCTCCAGTCACCCAGATTACCCGCTCCTGCAGGACGCCCTTCGTATCTCACAAAACTTCCTGTCCAGCATTAACGAAGAGAGCACTCCTCGCCGCCAGTCCATGACCGTGAAGAAAGGAGAG
VastDB Features
Vast-tools module Information
Secondary ID
ENSDARG00000079286:ENSDART00000101282:7
Average complexity
IR
Mappability confidence:
NA
Protein Impact
ORF disruption upon sequence inclusion
No structure available
Features
Disorder rate (Iupred):
C1=0.000 A=NA C2=0.191
Domain overlap (PFAM):
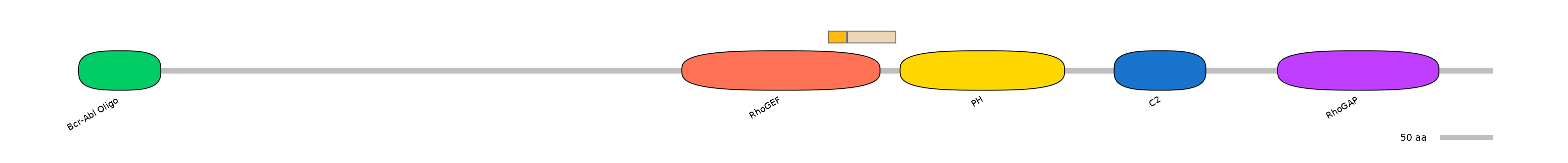
C1:
PF0062115=RhoGEF=FE(9.0=100)
A:
NA
C2:
PF0062115=RhoGEF=PD(16.4=66.0)
Main Inclusion Isoform:
NA
Other Inclusion Isoforms:
NA
Other Skipping Isoforms:
NA
Associated events
Conservation
Fruitfly
(dm6)
No conservation detected
Primers PCR
Suggestions for RT-PCR validation
F:
No suggested primer sequences
R:
No suggested primer sequences
Band lengths:
Functional annotations
There are 0 annotated functions for this event
GENOMIC CONTEXT[edit]
INCLUSION PATTERN[edit]